Page 121 - Revista 2019
P. 121
Marcadores de ancestría entre indígenas y mestizos. Criollo et al.
simultáneamente. ma para los 100 AIMs. Después de esto los datos fueron
guardados en formatos .ped y .map para ser usados en
Análisis estadísticos. Los datos genotípicos de los el programa PLINK, a los cuales se les realizó el control
AIMs se organizaron en dos archivos formato de texto de calidad para sacar del estudio aquellos individuos y
plano para el programa Plink, el .ped (ID familia, ID SNPs con más del 10% de datos genotípicos faltantes.
individuo, ID padre, ID madre, sexo y fenotipo en fi las Posterior al control de calidad, 36 (10,14%) individuos
y en columnas los genotipos) y el archivo .map (código fueron excluidos y se realizaron los análisis a 319 indi-
de los SNPs, cromosoma y posición). En Plink se reali- viduos.
zó el control de calidad de los datos genotípicos exclu-
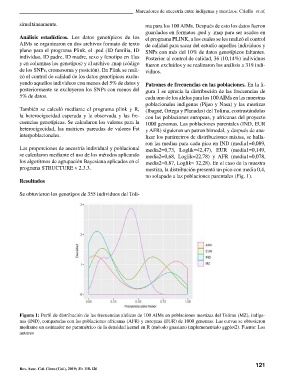
yendo aquellos individuos con menos del 5% de datos y Patrones de frecuencias en las poblaciones. En la fi -
posteriormente se excluyeron los SNPs con menos del gura 1 se aprecia la distribución de las frecuencias de
5% de datos. cada uno de los alelos para los 100 AIMs en las muestras
poblacionales indigenas (Pijao y Nasa) y las mestizas
También se calculó mediante el programa plink y R, (Ibagué, Ortega y Planadas) del Tolima, contrastándolas
la heterocigocidad esperada y la observada y las fre- con las poblaciones europeas, y africanas del proyecto
cuencias genotípicas. Se calcularon los valores para la 1000 genomas. Las poblaciones parentales (IND, EUR
heterocigocidad, las matrices pareadas de valores Fst y AFR) siguieron un patron bimodal, y después de ana-
interpoblacionales. lizar los parámetros de distribuciones mixtas, se halla-
ron las medias para cada pico en IND (media1=0,089,
Las proporciones de ancestría individual y poblacional media2=0,73, Loglik=42,47), EUR (media1=0,149,
se calcularon mediante el uso de los métodos aplicando media2=0,68, Loglik=22,78) y AFR (media1=0,078,
los algoritmos de agrupación Bayesiana aplicados en el media2=0,87, Loglik= 32,28). En el caso de la muestra
programa STRUCTURE v 2.3.3. mestiza, la distribución presentó un pico con media 0,4,
no solapado a las poblaciones parentales (Fig. 1).
Resultados
Se obtuvieron los genotipos de 355 individuos del Toli-
Figura 1: Perfi l de distribución de las frecuencias alélicas de 100 AIMs en poblaciones mestizas del Tolima (MZ), indíge-
nas (IND), comparadas con las poblaciones africanas (AFR) y europeas (EUR) de 1000 genomas. Las curvas se obtuvieron
mediante un estimador no paramétrico de la densidad kernel en R (método gausiano implemenentado ggplot2). Fuente: Los
autores
121
Rev. Asoc. Col. Cienc.(Col.), 2019; 31: 118-126